ในนิวเคลียสของเซลล์มนุษย์ (Homo sapiens sapiens) มีโครโมโซมทั้งหมดสองชุดที่คล้ายคลึงกัน (homologous chromosomes) แต่ละชุดมีโครโมโซมอยู่ 23 แท่ง บนโครโมโซมเหล่านี้มียีนที่ควบคุมลักษณะต่าง ๆ ทั้งหมดของ มนุษย์อยู่ประมาณ 25,000 ยีน ยึดเรียงกันอยู่อย่างเป็นลำดับ แล้วยีนเหล่านี้ยึดเรียงกันอยู่เป็นลำดับอย่างไร อยู่ใกล้ไกล กันเพียงใด อยู่บนโครโมโซมเดียวกันหรือไม่ คุณรู้ไหมว่ารังสีช่วยตอบคำถามเหล่านี้ได้
ลองคิดดูว่า หากเราสามารถหักโครโมโซมเป็นท่อนสั้น ๆ โดยที่ตำแหน่งต่าง ๆ ของโครโมโซมมีโอกาสที่จะถูกหักเท่า ๆ กัน จากนั้นเราจึงแยกท่อนโครโมโซมเหล่านั้นออกจากกัน โอกาสที่เราจะพบยีนสองยีนใด ๆ ซึ่งยึดเรียงอยู่ใกล้กันบน ท่อนโครโมโซมเดียวกัน มีมากกว่าโอกาสที่เราจะพบยีนสองยีนซึ่งอยู่ห่างกัน เพราะโอกาสที่เราจะหักโครโมโซม ณ ตำแหน่งระหว่างยีนสองยีนซึ่งอยู่ห่างกันมีมากกว่าโอกาสที่เราจะหักโครโมโซม ณ ตำแหน่งระหว่างยีนสองยีนซึ่งอยู่ ใกล้กัน เพียงใช้หลักการนี้ เราก็จะสามารถบอกได้ว่า ยีนสองยีนใด ๆ ยึดเรียงอยู่ใกล้หรือไกลจากกันบนโครโมโซม
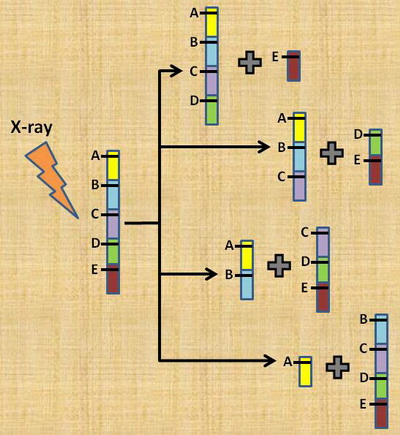
|
สมมติว่าเราแบ่งโครโมโซมออกเป็นห้าส่วน A ถึง E และหลังจากได้รับรังสี รอยต่อระหว่างส่วนทั้งห้านี้ มีโอกาสแตกหักได้เท่า ๆ กัน ดังนั้น เมื่อผ่านการฉายรังสี โอกาสที่จะพบส่วน A กับส่วน B บนชิ้นโครโมโซมเดียวกัน จึงมีมากกว่าโอกาสที่จะพบส่วน A กับส่วน C หรือส่วน A กับส่วน D หรือส่วน A กับส่วน E อยู่ด้วยกันบนชิ้นโครโมโซมหนึ่ง ๆ |
รังสีเข้ามามีบทบาทในการศึกษาการยึดเรียงของยีนโดยเฉพาะในเซลล์มนุษย์และเซลล์สัตว์ชั้นสูง เมื่อเราฉายรังสีเอกซ์ หรือแกมมาแก่เซลล์ในปริมาณที่สูงพอให้เซลล์ตาย (lethal dose) คือ 30-100 Gy รังสีจะทำให้โครโมโซมแตกหักและ ถูกทำลายมากจนเกินกว่าที่เซลล์จะสามารถซ่อมแซมได้ แต่เราไม่สามารถควบคุมขนาดของชิ้นโครโมโซม และตำแหน่ง ของการแตกหักได้ ชิ้นโครโมโซมที่ได้จึงมีขนาดใหญ่หรือเล็กแตกต่างกันไป และเกิดจากตำแหน่งของ การแตกหักต่าง ๆ กัน ในการศึกษาการยึดเรียงของยีน นักวิจัยใช้รังสีเอกซ์ร่วมกับการบังคับให้เซลล์รวมตัวกัน และการวิเคราะห์ ดีเอ็นเอเครื่องหมายเชิงสถิติ ในเทคนิคการทำแผนที่พันธุกรรมที่เรียกว่า Radiation Hybrid Mapping
ในการศึกษาการยึดเรียงยีนมนุษย์ก่อนยุคจีโนมิกส์ เซลล์มนุษย์จะถูกรวมตัวเข้าด้วยกันกับเซลล์หนูให้เป็นเซลล์เดียวกัน ผ่านกระบวนการรวมเซลล์ (cell fusion) โดยอาศัยสารเคมี polyethylene glycol หรืออาศัยไวรัสบางชนิด เซลล์ที่ รวมตัวเข้าด้วยกันในเบื้องต้นจะยังคงนิวเคลียสจากทั้งสองสปีชีส์ไว้ เรียกเซลล์ที่มีลักษณะเช่นนี้ว่าเฮเทอโรแคริออน (heterokaryon) เมื่อมีการแบ่งเซลล์ เซลล์ใหม่ที่ได้จะมีเพียงนิวเคลียสเดียวเรียกเซลล์เหล่านี้ว่าเซลล์ลูกผสมหนู-มนุษย์ (rodent-human hybrid cells) ในเบื้องต้นเซลล์เหล่านี้จะยังไม่เสถียร เนื่องจากโครโมโซมของคนไม่สามารถ จำลองตัวเองได้ดี เมื่อเกิดการแบ่งเซลล์จึงมักเกิดการสูญหายของโครโมโซมคนโดยที่โครโมโซมของหนูยังอยู่ครบ เซลล์ลูกผสมที่เสถียรจะมีโครโมโซมจากคนเหลืออยู่เพียง 3 - 4 โครโมโซมเท่านั้น
เราสามารถรวบรวมเซลล์ลูกผสมที่มีโครโมโซมต่าง ๆ ของคนไว้ด้วยกันเพื่อศึกษาตำแหน่งของยีนบนโครโมโซม จัดเป็นชุดเซลล์ลูกผสม (hybrid cell panel) และหากเรามีดีเอ็นเอเครื่องหมาย (DNA markers) ของยีนที่เราสนใจ ไม่ว่าจะอยู่ในรูปแบบ PCR primers หรือ hybridization probe เราก็จะสามารถตรวจสอบได้ทันทีว่ายีนของเราอยู่บน โครโมโซมใด โดยใช้ชุดเซลล์ลูกผสมดังกล่าว
อย่างไรก็ตาม การประยุกต์ใช้ชุดเซลล์ลูกผสมในรูปแบบนี้ ทำให้เราทราบแต่เพียงว่ายีนต่าง ๆ อาศัยอยู่บนโครโมโซมใด แต่ไม่สามารถบอกเราได้ว่ายีนเหล่านั้นยึดเรียงกันเป็นลำดับอย่างไรในแต่ละโครโมโซม นักวิจัยจึงต้องสร้างชุดเซลล์ ลูกผสมอันประกอบด้วยเซลล์ลูกผสมที่ได้รับโครโมโซมมนุษย์เพียงโครโมโซมเดียว (monochromosomal hybrids) เมื่อนำไปฉายรังสีที่ปริมาณรังสีถึงตาย ซึ่งทำให้โครโมโซมทั้งของมนุษย์และหนูแตกหักมากเกินกว่าจะซ่อมแซมได้ เซลล์ลูกผสมนั้นจะไม่สามารถดำรงชีพอยู่ต่อไป ในการทำแผนที่พันธุกรรม นักวิจัยจำเป็นต้องเก็บรักษาชิ้นโครโมโซม เหล่านี้ไว้ศึกษา จึงนำเซลล์ที่ผ่านการฉายรังสีนั้นมารวมตัวเข้าเซลล์หนูปกติอีกครั้ง เกิดเป็นเซลล์ลูกผสมผู้ให้-ผู้รับ (donor-recipient hybrid cells) โดยมีเซลล์ monochromosomal hybrid ที่ผ่านการฉายรังสีเป็นเซลล์ผู้ให้ (donor) และเซลล์หนูปกติเป็นเซลล์ผู้รับ (recipient)
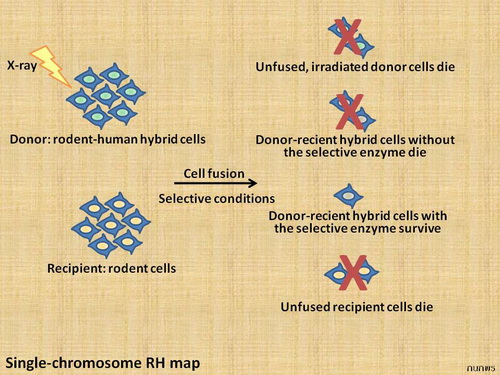
|
นักวิจัยใช้เอนไซม์ที่เซลล์ผู้ให้ผลิตได้ แต่เซลล์ผู้รับผลิตไม่ได้ (selective enzyme) ในการคัดเลือกเซลล์ลูกผสม ที่ได้รับชิ้นโครโมโซมจากเซลล์ผู้ให้ |
เนื่องจากการฉายรังสีอาจทำลายส่วนของโครโมโซมที่สำคัญต่อการถ่ายทอดโครโมโซมผ่านการแบ่งเซลล์ เช่น เซนโทรเมียร์ (centromere) และเทโลเมียร์ (telomere) นักวิจัยจึงต้องระบุลักษณะเฉพาะของเซลล์ผู้ให้และเซลล์ผู้รับ และคัดเลือกเซลล์ลูกผสมผู้ให้-ผู้รับ ที่มีลักษณะจำเพาะ เซลล์ผู้รับที่นิยมใช้มักขาดความสามารถในการผลิตเอนไซม์ ที่จำเป็นต่อการดำรงชีพในอาหารเลี้ยงเซลล์สำหรับคัดเลือก (selection medium) เช่น เอนไซม์ hypoxanthine guanine phosphoribosil transferase และ thymidine kinase และเซลล์ผู้ให้ต้องเป็นเซลล์ที่สามารถผลิตเอนไซม์ นี้ได้ โดยมียีนสำหรับเอนไซม์นี้อยู่บนโครโมโซมใดโครโมโซมหนึ่ง จึงเรียกยีนนั้นว่าเป็นเครื่องหมายสำหรับ การคัดเลือก (selection marker) เมื่อนำเซลล์ผู้ให้และเซลล์ผู้รับ มารวมตัวกัน เซลล์ลูกผสมที่ได้จะถูกเลี้ยงบนอาหาร คัดเลือก ดังนั้น เซลล์ลูกผสมที่ได้รับยีนสำหรับเอนไซม์นี้ บนชิ้นโครโมโซมที่สามารถถ่ายทอดได้เท่านั้น จึงจะมีชีวิต อยู่รอดและ ขยายพันธุ์ได้บนอาหารคัดเลือก หากชิ้นโครโมโซมที่มียีนสำหรับการคัดเลือก ขาดเซนโทรเมียร์หรือ เทโลเมียร์ มันจะต้องต่อเข้ากับชิ้นโครโมโซมอื่น ที่มีส่วนสำคัญของโครโมโซมดังกล่าว จึงจะสามารถถ่ายทอดได้ทางพันธุกรรม
เซลล์ลูกผสมผู้ให้-ผู้รับจะถูกแยกเลี้ยงเป็นโคลนเซลล์ลูกผสม (hybrid cell clones) โดยแต่ละโคลนมีโอกาส ที่จะได้รับ ชิ้นส่วนของโครโมโซมมนุษย์ที่แตกต่างกันออกไป ชุดของเซลล์ลูกผสมเช่นนี้ จะถูกนำไปใช้เพื่อศึกษาการยึดเรียงของ ยีนมนุษย์ต่อไป โดย ชุดเซลล์ลูกผสมที่มีขนาดประมาณ 40 50 โคลน อาจเพียงพอสำหรับการศึกษาการยึดเรียงของ กลุ่มยีนกลุ่มหนึ่งบนโครโมโซม
ในการศึกษาดังกล่าวเซลล์ลูกผสมผู้ให้-ผู้รับ จะถูกตรวจสอบด้วยดีเอ็นเอเครื่องหมาย ความถี่ของการตรวจพบ ดีเอ็นเครื่องหมายสองเครื่องหมายใด ๆ ในแต่ละโคลนของชุดเซลล์ลูกผสมจะถูกนำมาคำนวณเชิงสถิติ เพื่อให้ได้มา ซึ่งระยะห่างระหว่างตำแหน่งของดีเอ็นเครื่องหมายนั้นบนโครโมโซม ซึ่งข้อมูลนี้จะช่วยให้นักวิจัยสามารถวิเคราะห์หา ลำดับการยึดเรียงของยีนและดีเอ็นเอเครื่องหมายเหล่านั้นได้ในที่สุด ลำดับและกลุ่มการยึดเรียงของยีนหรือ ดีเอ็นเอเครื่องหมายที่ได้จากเทคนิคนี้เรียกว่า radiation hybrid map (RH map)
ในปัจจุบันเทคนิคนี้ได้รับการขยายขอบเขตเพื่อศึกษาโครโมโซมทั้งหมดในคราวเดียว (whole genome studies) เพราะ อาศัยขั้นตอนวิธีเชิงคอมพิวเตอร์ (computer algorithm) สำหรับวิเคราะห์ข้อมูล และเทคโนโลยีดีเอ็นเอเครื่องหมาย (DNA markers) ได้รับการพัฒนาอย่างมาก ช่วยให้สามารถแยกแยะกลุ่มการยึดเรียง (linkage group) ต่าง ๆ ได้แม้จะมี ข้อมูลและกลุ่มการยึดเรียงเป็นจำนวนมาก ปัจจุบันจึงใช้เซลล์มนุษย์ซึ่งไม่ได้ผ่านการรวมตัวกับเซลล์หนูมาก่อน เป็นเซลล์ผู้ให้ที่จะต้องผ่านการฉายรังสี โดยเซลล์ผู้รับยังเป็นเซลล์หนูเช่นเดิม หลังจากกระบวนการรวมเซลล์ ชิ้นส่วน ของโครโมโซมของคนบางชิ้น จะเข้าแทนที่ชิ้นส่วนโครโมโซมของหนูที่มีลำดับดีเอ็นเอคล้ายคลึงกัน โดยผ่าน กระบวนการแลกเปลี่ยนชิ้นส่วนของโครโมโซมแบบ homologous recombination หรือใช้กระบวนการซ่อมแซม ดีเอ็นเอ เช่น non-homologous end joining โดยแต่ละเซลล์ลูกผสมอาจได้รับชิ้นส่วนจากโครโมโซมต่าง ๆ มากกว่าหนึ่ง โครโมโซม และชิ้นโครโมโซมเหล่านี้จะแตกต่างกันในแต่ละเซลล์ลูกผสม ดังนั้น ในการวิเคราะห์พันธุกรรม ทั้งหมด ในการทดลองชุดเดียว จะต้องใช้ชุดเซลล์ลูกผสม ที่มีขนาดประมาณ 100-200 โคลน เป็นอย่างน้อย
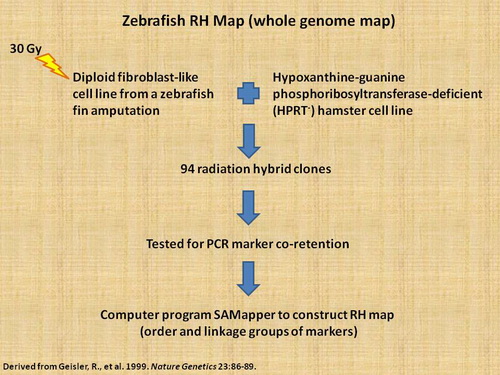
|
ตัวอย่างขั้นตอนการทำ RH map สำหรับปลา zebrafish (Danio rario) โดยใช้ชุดเซลล์ลูกผสม zebrafish hamster
|
นอกจากการยึดเรียงของยีนในเซลล์มนุษย์ ปัจจุบันเทคนิคนี้ยังเป็นที่นิยมใช้กับเซลล์สัตว์ เช่น วัว หมู ไก่ และปลา อีกด้วย สำหรับพืชไม่นิยมใช้เทคนิคนี้ เนื่องจากการเพาะเลี้ยงเซลล์พืชมีข้อจำกัดที่แตกต่างจากการเพาะเลี้ยงเซลล์สัตว์ และผู้วิจัยก็ยังสามารถทำการผสมพันธุ์และปลูกเลี้ยงพืชต่าง ๆ เพื่อวิเคราะห์การยึดเรียงของยีนได้ พืชที่มีการทำ RH map เช่น ข้าวโพด และข้าวสาลี เป็นต้น
เอกสารเพื่อศึกษาเพิ่มเติม
- Alberts, B. et al. 2002. Molecular Biology of the Cells. 4 th edition. New York; Garland Science.
- Cox, D.R., et al. 1990. Radiation Hybrid Mapping: A Somatic Cell Genetic Method for Constructing High-Resolution Maps of Mammalian Chromosomes. Science 250: 245250.
- Geisler, R.., et al. 1999. A radiation hybrid map of the zebrafish genome. Nature Genetics 23:86-89.
- Klein, C . Radiation Hybrid Mapping . (On line) http://www.biologyreference.com/Po-Re/Radiation-Hybrid-Mapping.html.
- Riera-Lizarazu, O., M.I. Vales, and S.F. Kianian. 2008. Radiation hybrid (RH) and HAPPY mapping in plants. Cytogenet. Genome Res. 120: 233-240.
- Strachen, T., and A.P. Read. 1999. Human Molecular Genetics. 2 nd edition. New York; Wiley-Liss.
|